En un nuevo estudio, los investigadores de IBM, Oxford University y de Diamond Light Source muestran que el modelo de Inteligencia Artificial de IBM, MoLFormer, puede generar moléculas antivirales para múltiples proteínas virales objetivo, que pueden acelerar el proceso de descubrimiento de fármacos.
Los investigadores de la Oxford University validaron con éxito las propiedades antivirales de once moléculas.
Este avance tiene el potencial de llevar medicamentos a las personas más rápido en la próxima crisis y poner al alcance de la mano tratamientos para enfermedades urgentes que amenazan la vida.
IA para diseñar moléculas
Al inicio de la pandemia del COVID-19, un grupo de científicos informáticos de IBM quería explorar si la Inteligencia Artificial generativa podría usarse para diseñar moléculas nunca antes vistas para bloquear el SARS-CoV-2, el virus que causa el COVID-19.
David Stuart, jefe de la Division of Structural Biology en el Department of Clinical Medicine en la University of Oxford y director de ciencias para la vida en Diamond Light Source, el sincrotrón nacional del Reino Unido que es una autoridad en patógenos VIH, SARS y Ébola, entre otros virus explica que inicialmente se mostró escéptico.
“La idea de que se pudiera tomar una secuencia de proteínas y, con IA, sacar de la nada sustancias químicas que se unirían a un sitio 3D en el virus parecía muy poco probable”, manifestó el científico.
Sin embargo, él y Martin Walsh, biólogo estructural experto y director adjunto de ciencias de la vida en Diamond, se unieron al equipo de IBM y, en el transcurso de tres años, demostraron que la IA generativa podría “sacar puntos de partida viables para los antivirales de la nada”.
Debido a que el modelo generativo también era un modelo básico, entrenado previamente en cantidades masivas de datos sin procesar, era lo suficientemente versátil como para crear nuevos inhibidores para múltiples objetivos de proteínas sin capacitación adicional ni conocimiento de su estructura 3D.
Los grupos de Stuart y Walsh habían comenzado a trabajar en dos proteínas esenciales del SARS-CoV-2, a saber, la proteína espiga y la proteasa principal.
Con estos objetivos, el equipo encontró cuatro posibles antivirales contra el COVID-19 en una fracción del tiempo que habría tomado con los métodos convencionales.
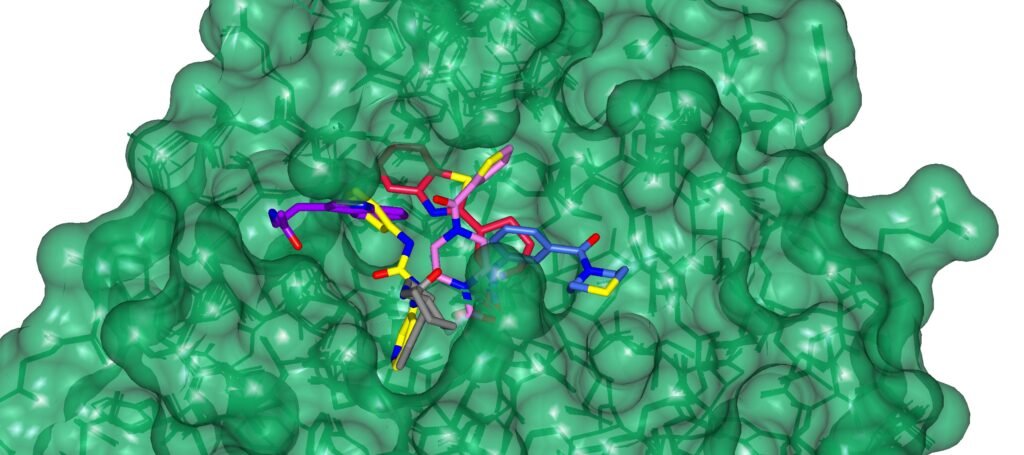
Luego, el estudio aprovechó las líneas de luz de cristalografía macromolecular de alto rendimiento de Diamond para visualizar cómo un subconjunto de los compuestos generados por IA se unía a la proteasa principal.
El equipo afirmó que las moléculas validadas tienen muchos más obstáculos que superar, incluidos los ensayos clínicos, antes de que las empresas puedan potencialmente convertirlas en medicamentos.
Pero incluso si los “éxitos” generados por la inteligencia artificial nunca se materializan en medicamentos reales, el estudio confirma que la IA generativa tiene un papel importante que desempeñar en el futuro del desarrollo de medicamentos, especialmente en tiempos de crisis.
“Tomó tiempo desarrollar y validar estos métodos, pero ahora que tenemos una tubería de trabajo, podemos generar resultados mucho más rápido”, dijo el coautor principal del estudio, Payel Das, investigador en IBM Research. “Cuando surja el próximo virus, la IA generativa podría ser fundamental en la búsqueda de nuevos tratamientos”.
“La generación de compuestos iniciales que se unen con alta afinidad a un objetivo farmacológico de interés acelera el descubrimiento de fármacos basados en la estructura y respalda nuestros esfuerzos para estar mejor preparados para futuras pandemias”, dijo Martin Walsh, otro de los coautores principales.
Controlled Generation of Molecules – CogMol
Los científicos construyeron sus modelo “Controlled Generation of Molecules – CogMol”, en una arquitectura de IA generativa conocida como codificadores automáticos variacionales (VAE).
Los VAE codifican los datos sin procesar en una representación comprimida y luego los decodifican o traducen nuevamente a una variación estadística de la muestra original.
El modelo fue entrenado en una conjunto de datos de moléculas representadas como cadenas de texto, junto con información general sobre proteínas y sus propiedades de unión. Pero deliberadamente omitieron información sobre la estructura 3D del SARS-CoV-2 o las moléculas que se conoce se unen a el.
El objetivo era dar a su modelo base generativo una amplia base de conocimiento para que pudiera implementarse más fácilmente para tareas de diseño molecular que nunca antes había visto.
La finalidad era encontrar moléculas similares a fármacos que se unieran a dos objetivos proteicos del COVID: la espiga, que transmite el virus a la células huésped, y la proteasa principal que ayuda a propagarlo.
Aunque las estructuras 3D de ambas proteínas ya se habían descubierto en ese momento, los investigadores de IBM optaron por usar solo sus secuencias de aminoácidos, derivadas de su ADN. Al limitarse de esta manera, esperaban que el modelo pudiera aprender a generar moléculas sin conocer la forma de su objetivo.
Los investigadores ingresaron solo la secuencia de aminoácidos para cada proteína objetivo en CogMol, que generó 875,000 moléculas candidatas en tres días.
Para reducir el número de moléculas, los científicos analizaron a los candidatos a través de una plataforma de retrosíntesis (IBM RXN for Chemistry), para comprender qué ingredientes se necesitarían para sintetizar los compuestos.
De acuerdo con las recetas predichas de la plataforma, los científicos seleccionaron 100 moléculas para cada objetivo. Los químicos de Enamine redujeron aún más la lista a cuatro moléculas para cada objetivo, seleccionando las que se consideraban más fáciles de fabricar.
Evaluación de las nuevas moléculas
Después de sintetizar las ocho nuevas moléculas, Enamine las envió a Oxford para probar su capacidad para interrumpir las funciones de los dos objetivos proteicos en los laboratorios del profesor Chris Schofield y el Profesor Gavin Screaton.
El intenso haz de rayos X generado por Diamond, que es 10 mil millones de veces más brillante que el sol, se utilizó para visualizar cómo los compuestos interactuaban con las proteínas para inactivar su función.
Los nuevos compuestos se probaron además en pruebas de inhibición de objetivos y de neutralización de virus vivos.
Dos de los antivirales validados se dirigieron a la proteasa principal; los otros dos no solo se dirigieron a la proteína espiga, sino que demostraron ser capaces de neutralizar las seis variantes principales del COVID.
“Obtienes un mapa que muestra exactamente dónde se unen las cosas y ¡bang! tienes una confirmación”, destacó Stuart.
CogMol es uno de varios modelos de base química que IBM ha desarrollado desde entonces. El más grande, MolFormer-XL, se entrenó en una base de datos de más de 1.100 millones de moléculas y Moderna lo está utilizando actualmente para diseñar medicamentos de ARNm.
“Creamos puntos de partida válidos para el desarrollo acelerado de antivirales utilizando un modelo de base generativo que sabía relativamente poco sobre sus objetivos proteicos”, describe el coautor principal del estudio, Jason Crain, un investigador de IBM Research y profesor en Oxford.
«Tengo la esperanza de que estos métodos nos permitan crear antivirales y otros compuestos que se necesitan con urgencia mucho más rápido y de forma más económica en el futuro», dijo Crain.
Aunque los investigadores se centraron en validar los antivirales para covid, argumentan que estos métodos pueden extenderse a virus existentes que continúan mutando, como la gripe, o virus que aún no han aparecido.
“Si quiere estar preparado para la próxima pandemia, quiere medicamentos que actúen en diferentes sitios de la proteína”, concluyó Stuart. “Se vuelve mucho más difícil para el virus escapar”.
Puedes descargar el modelo de Inteligencia Artificial de IBM, MoLFormer.
Referencia (acceso abierto):
Vijil Chenthamarakshan, Samuel C. Hoffman , C. David Owen, Petra Lukacik, Claire Strain-Damerell Daren Fearon ,Tika R. Malla, Anthony Tumber, Christopher J. Schofield, Helen M.E. Duyvesteyn, Wanwisa Dejnirattisai, Loic Carrique, Thomas S. Walter, Gavin R. Screaton, Tetiana Matviiuk, Aleksandra Mojsilovic, Jason Crain, Martin A. Walsh, David I. Stuart , and Payel Das. 2023. Accelerating drug target inhibitor discovery with a deep generative foundation model.Sci. Adv.9,eadg7865(2023). DOI:10.1126/sciadv.adg7865
Editor y fundador de «Innovar o Morir». Milthon es Máster en Gestión de la Ciencia y la Innovación por la Universidad Politécnica de Valencia, con diplomas de especialización en Innovación Empresarial (UPV) y Gestión de la Innovación Orientada al Mercado (UPCH-Universitat Leipzig). Cuenta con experiencia práctica en la gestión de la innovación, habiendo liderado la Unidad de Innovación en Pesca del Programa Nacional de Innovación en Pesca y Acuicultura (PNIPA) y trabajado como consultor en diagnóstico para innovación abierta y vigilancia tecnológica. Cree firmemente en el poder de la innovación y la creatividad como motores de cambio y desarrollo.